今天介绍一些WSI分类的文献。涉及三种比较主要的技术:传统的特征分类,CNN和Transformer。
第二次更新
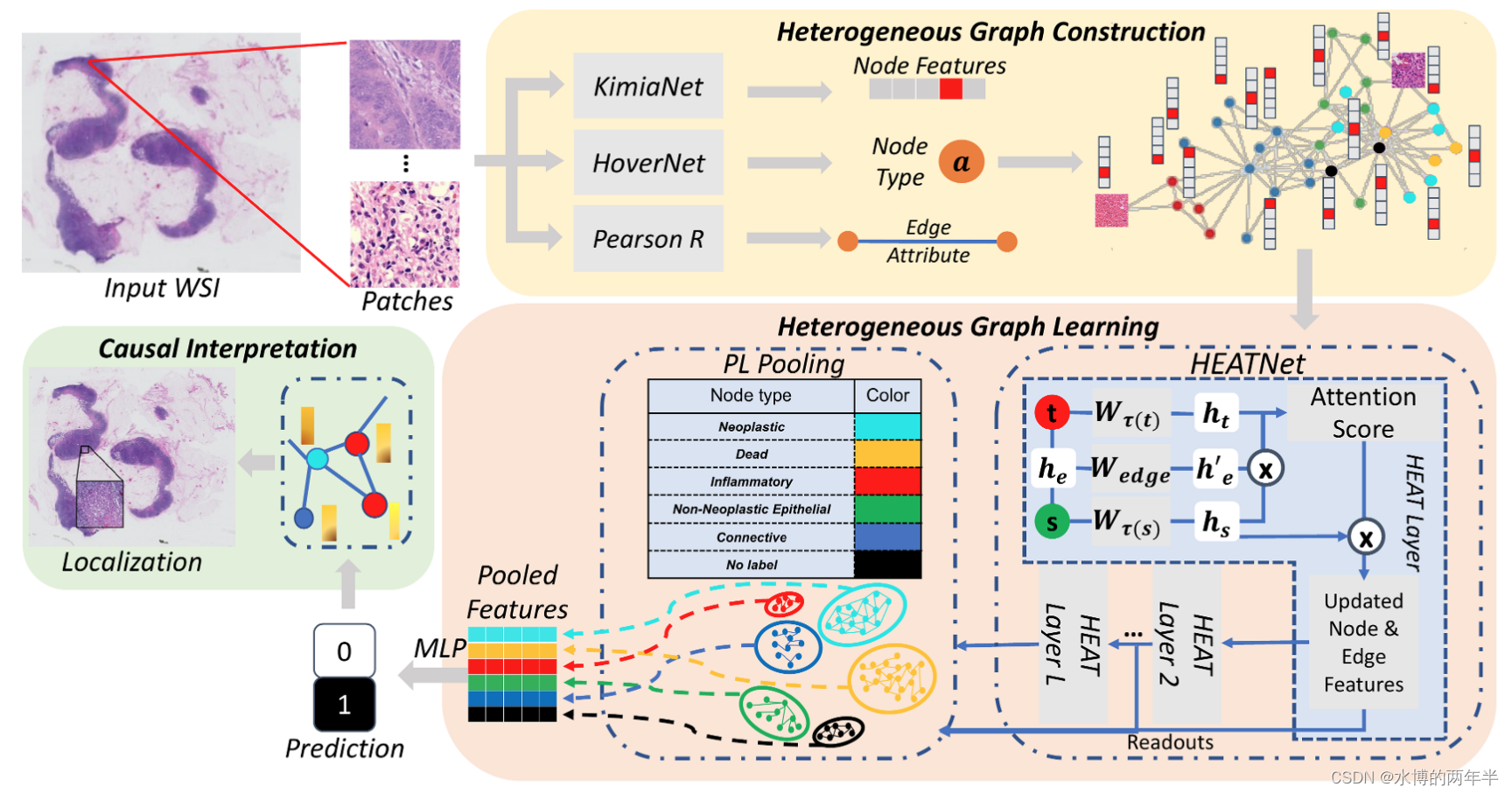
Histopathology Whole Slide Image Analysis With Heterogeneous Graph Representation Learning(CVPR2023)
我们设计了一种新的基于伪标签的语义一致池化机制来获取图级特征,从而缓解了传统的基于聚类池化的过度参数化问题。此外,观察到现有的基于关联的定位方法的局限性,我们提出了一种因果驱动的方法来归因于每个节点的贡献,以提高框架的可解释性。
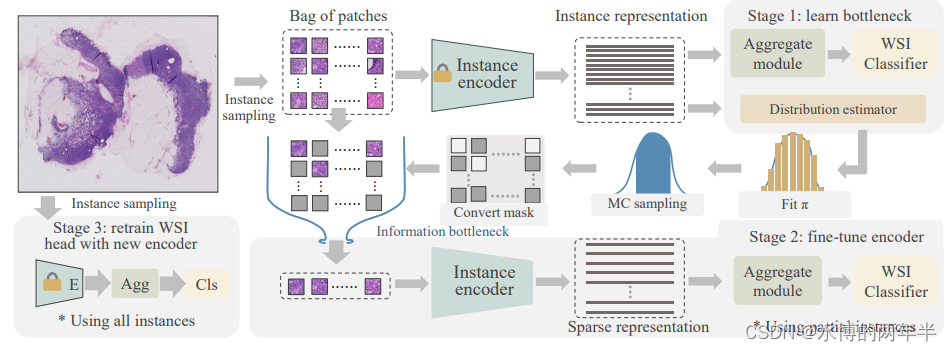
Task-specific Fine-tuning via Variational Information Bottleneck for Weakly-supervised Pathology Whole Slide Image Classification (cvpr2023)
我们提出了一种基于信息瓶颈理论的高效WSI微调框架。该理论使框架能够找到WSI的最小充分统计量,从而支持我们仅依赖于WSI级弱标签将骨干调整为特定于任务的表示。进一步分析WSI-MIL问题,从理论上推导出我们的微调方法。
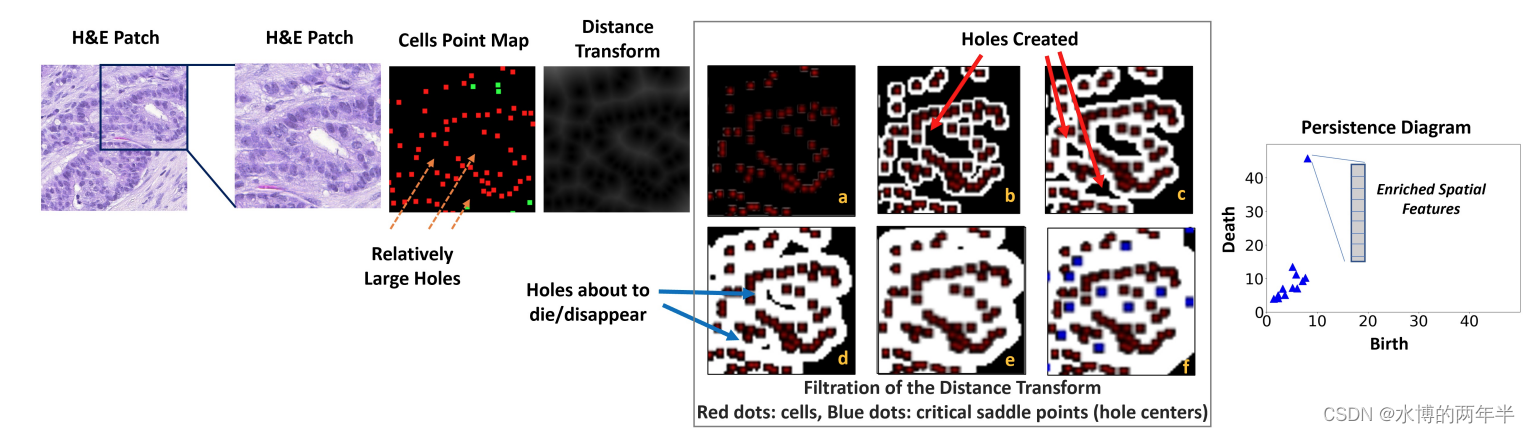
Topology-Guided Multi-Class Cell Context Generation for Digital Pathology(cvpr2023, 不是分类,但也挺好玩)
在数字病理学中,细胞的空间背景对细胞分类、癌症诊断和预后很重要。然而,对如此复杂的细胞环境进行建模是具有挑战性的。细胞形成不同的混合物、谱系、簇和孔。为了以可学习的方式对这种结构模式建模,我们从空间统计和拓扑数据分析中引入了几个数学工具。我们将这种结构描述符作为条件输入和可微损失合并到深度生成模型中。通过这种方式,我们第一次能够生成高质量的多类单布局。我们表明,拓扑丰富的单布局可用于数据增强,并提高下游任务(如单分类)的性能。
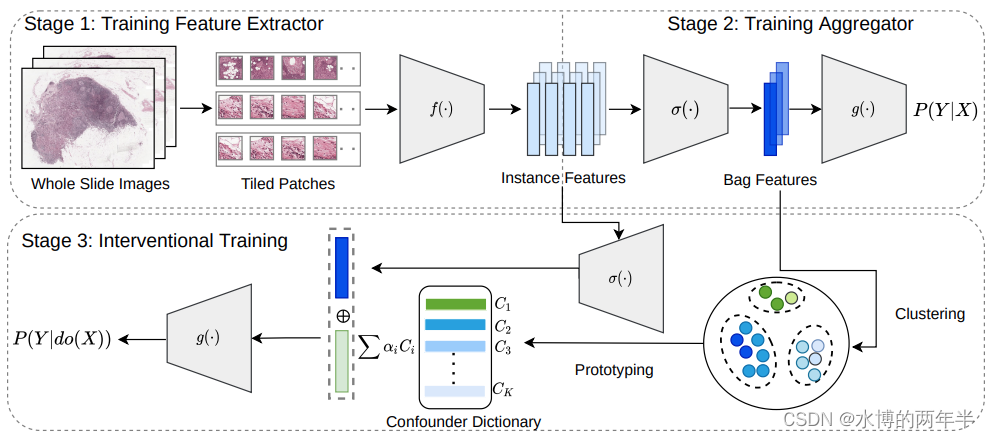
Interventional Bag Multi-Instance Learning On Whole-Slide Pathological Images (cvpr2023)
在本文中,我们提出了一种新的方案,介入袋多实例学习(IBMIL),以实现去发现袋级预测。与传统的基于似然的策略不同,本文提出的方案是基于后门调整来实现介入训练,从而能够抑制由袋上下文先验引起的偏差。请注意,IBMIL的原理与现有的袋MIL方法是正交的。因此,ibm能够为现有方案带来一致的性能提升,实现新的最先进的性能。
第一次更新:
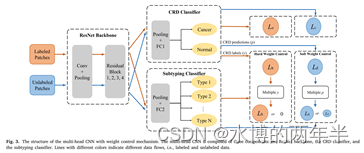
A semi-supervised multi-task learning framework for cancer classification with weak annotation in whole-slide images
在WSI中将非癌变区域视为癌变区域,这在训练过程中混淆了分型模型。针对后者的局限性,之前的研究提出先进行CRD,排除非癌变区域,然后基于剩余癌变斑块训练亚型模型。然而,单独训练忽略了这两个任务的交互作用,也会导致将CRD任务的错误传播给子类型任务。为了解决这些问题并同时提高CRD和亚型任务的性能,我们提出了一种用于癌症分类的半监督多任务学习(MTL)框架。我们的框架由一个主干特征提取器、两个特定于任务的分类器和一个权重控制机制组成。骨干特征提取器由两个特定于任务的分类器共享,这样就可以捕获CRD和子类型任务之间的交互。权重控制机制保留了这两个任务之间的顺序关系,保证了MTL框架下从子类型任务到CRD任务的错误反向传播。
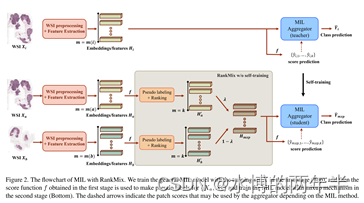
RankMix: Data Augmentation for Weakly Supervised Learning of Classifying Whole Slide Images with Diverse Sizes and Imbalanced Categories (CVPR2023)
我们提出了RankMix,这是一种在一对wsi中混合排名特征的数据增强方法。RankMix引入了伪标记和排序的概念,以便提取关键的WSI区域,为WSI分类任务做出贡献。进一步提出了一种两阶段的训练方法,以提高训练的稳定性和模型性能。
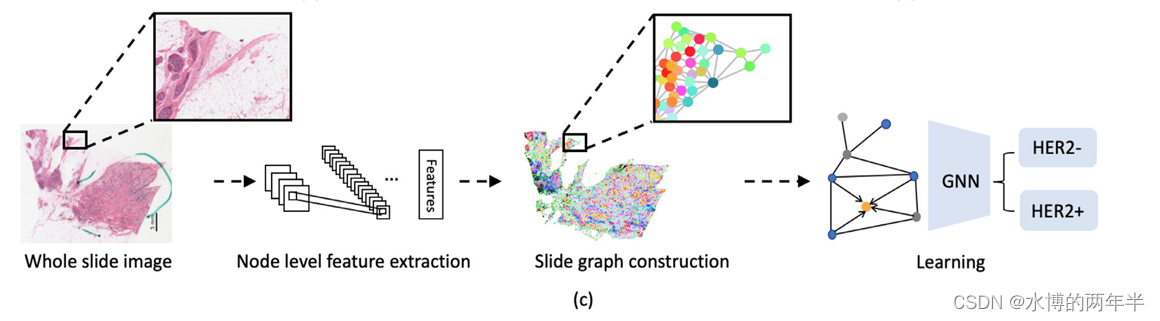
SlideGraph + : Whole slide image level graphs to predict HER2 status in breast cancer (MIA 2022)
我们提出了一种新的基于图神经网络(GNN)的模型(Slide- graph +),直接从常规血红素和伊红(H&E)染色的载玻片的整片图像预测HER2状态。
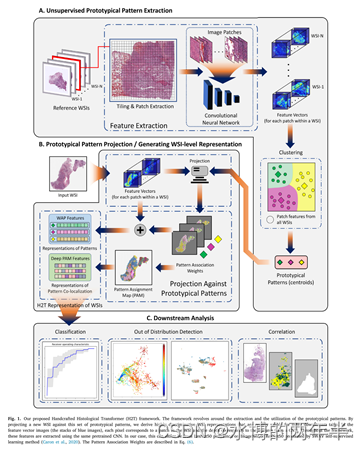
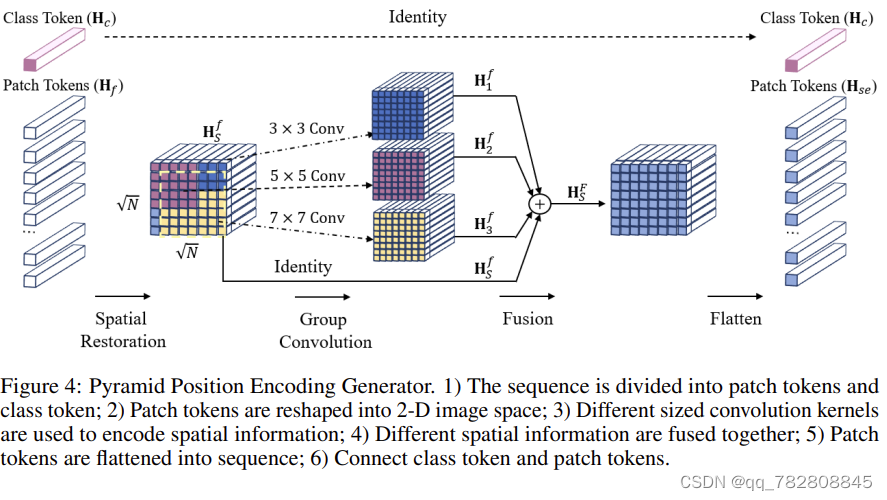
Handcrafted Histological Transformer (H2T): Unsupervised representation of whole slide images (MIA 2023)
我们提出了一个基于深度CNN的手工框架,用于构建整体的wsi级表示。基于最近在自然语言处理领域关于Transformer内部工作的发现,我们将其过程分解并将其手工制作成一个更透明的框架,我们称之为手工制作组织学Transformer或H2T。
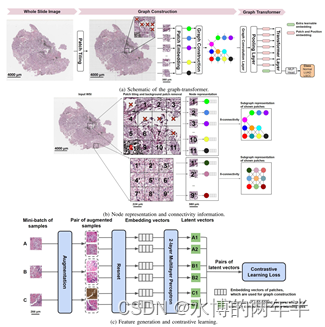
A Graph-Transformer for Whole Slide Image Classification (TMI 2022)
在执行监督深度学习时,将WSI划分为小块,训练并汇总结果以估计疾病等级。然而,基于patch的方法在训练过程中引入了标签噪声,因为它假设每个patch都是独立的,与WSI具有相同的标签,并且忽略了在疾病分级中重要的整体WSI级别信息。1、我们提出了一种图转换器(GT),它融合了基于图的WSI表示和用于处理病理图像的视觉转换器,称为GTP,以预测疾病等级。2、我们还介绍了一种基于图的显著性映射技术,称为GraphCAM,它可以识别与类标签高度相关的区域。
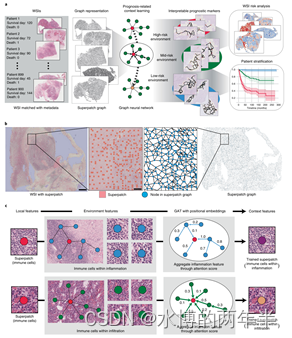
Derivation of prognostic contextual histopathological features from whole-slide images of tumours via graph deep learning (NBE 2022)
Transformer-based unsupervised contrastive learning for histopathological
image classification (MIA 2022)
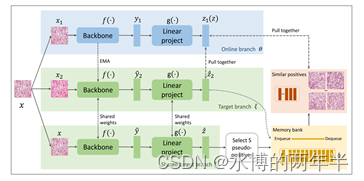
我们提出了一种新的自监督学习策略,称为语义相关对比学习(SRCL),它比较实例之间的相关性以挖掘更多的正对。与传统对比学习中来自一个实例的两个视图相比,我们的SRCL将多个积极实例与相似的视觉概念对齐,这增加了积极实例的多样性,然后产生更多信息的表征。我们采用混合模型(CTransPath)作为主干,该模型是通过集成卷积神经网络(CNN)和多尺度Swin Transformer架构而设计的。CTransPath在大量未标记的组织病理图像上进行预训练,可以作为协作的局部-全局特征提取器来学习更适合组织病理图像领域任务的通用特征表示。
传统特征WSI分类
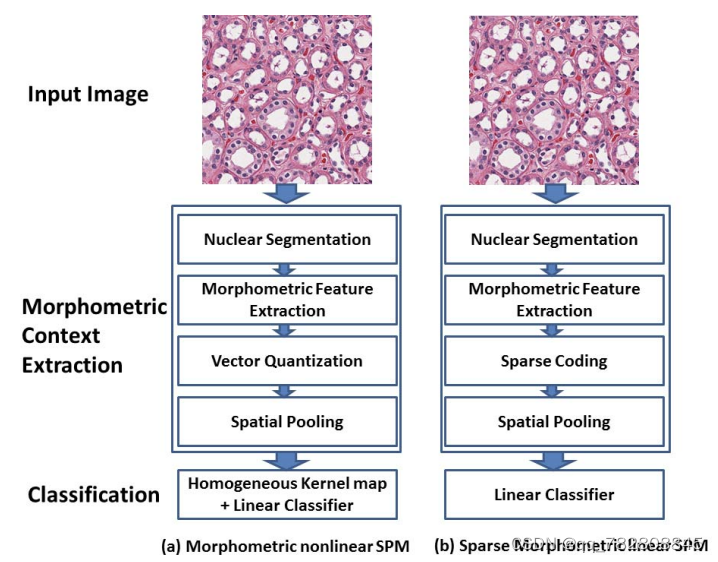
Classification of Tumor Histology via Morphometric Context
这个比较简单,主要利用细胞核的形态对WSI进行分类。用的是TCGA数据集。
Predicting non-small cell lung cancer prognosis by fully automated microscopic pathology image features
这个是NC的文章,很像影像组学,就是提取一堆特征,然后建模预后。评估非小细胞肺癌。
基于CNN的分类方法
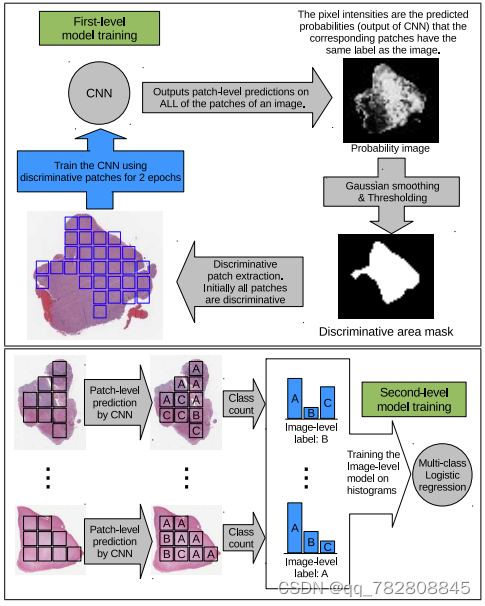
Patch-based Convolutional Neural Network for Whole Slide Tissue Image Classification
这是CVPR的文章。
首先是对每个patch进行训练,然后将训练的预测结果构建一个灰度直方图,然后再用直方图训练一个分类模型。
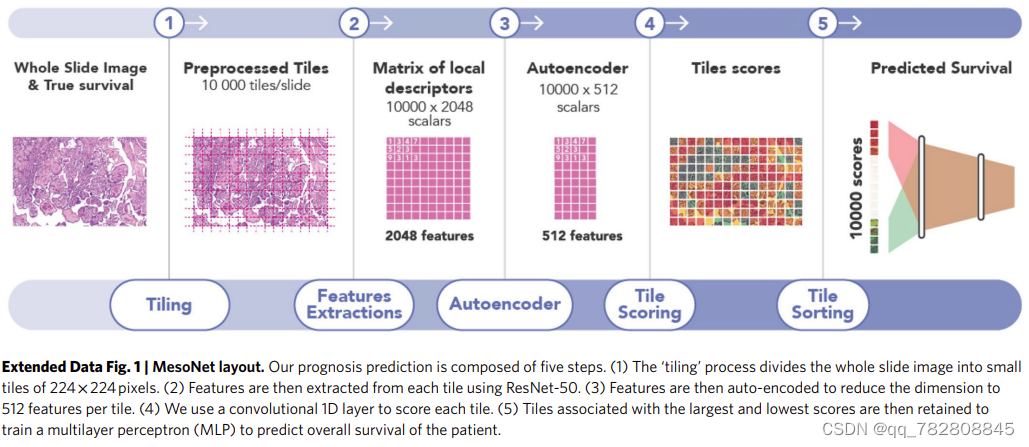
Deep learning-based classification of mesothelioma improves prediction of patient outcom
这个是nature medicine的文章。
使用的也是对patch提取特征,然后构建特征矩阵,接着用CNN预测的路线。这种方法做的人还是比较少了。
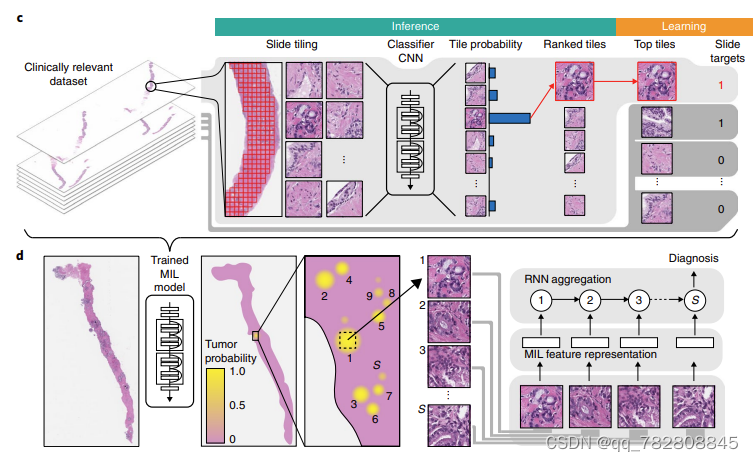
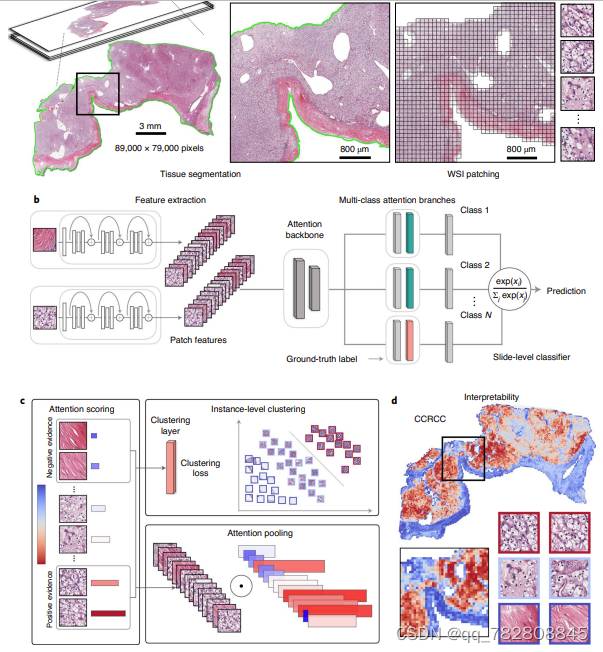
Clinical-grade computational pathology using weakly supervised deep learning on whole slide images
这个也是nature medicine的文章,算是比较令人熟知的WSI分类文章之一。
原理也很简单,就是用CNN选择出概率值比较高的patch,然后使用RNN分类。效果很不错,从文中的报道看。
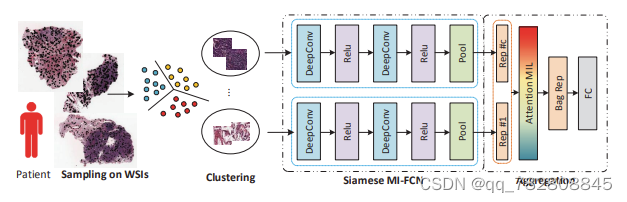
Whole slide images based cancer survival prediction using attention guided deep multiple instance learning networks
MIA的文章,算是WSI分类做的比较早的MIA了。
他的想法比较有意思。整体上是多实例学习MIL。但他将多实例细化了。首先用无监督对patch进行聚类,然后将每一个类当成是一个包。也比较make sense.因为病理图像中可能多种不同的组织,聚类可以让组织的patch大致的聚集到一块。
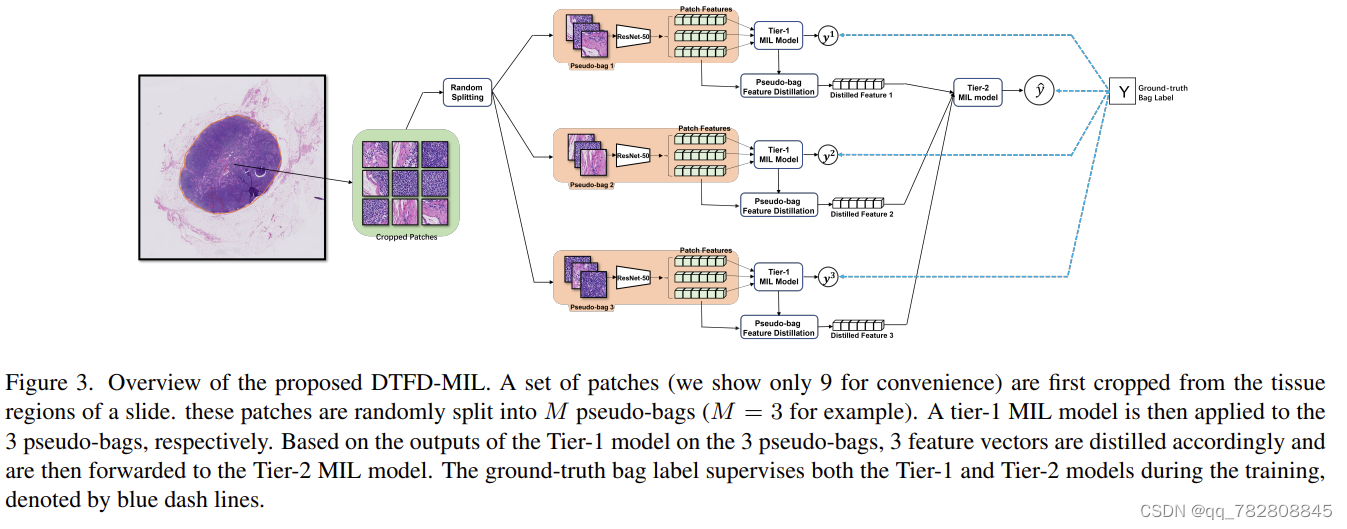
DTFD-MIL: Double-Tier Feature Distillation Multiple Instance Learning for Histopathology Whole Slide Image Classification
这是CVPR2022的文章。
和上面的文章类似,也是分成多个包处理。但他的伪包是随机分配的。出发点就是WSI中的patch太多,一个包学不到足够的知识。所以分级处理多个包。
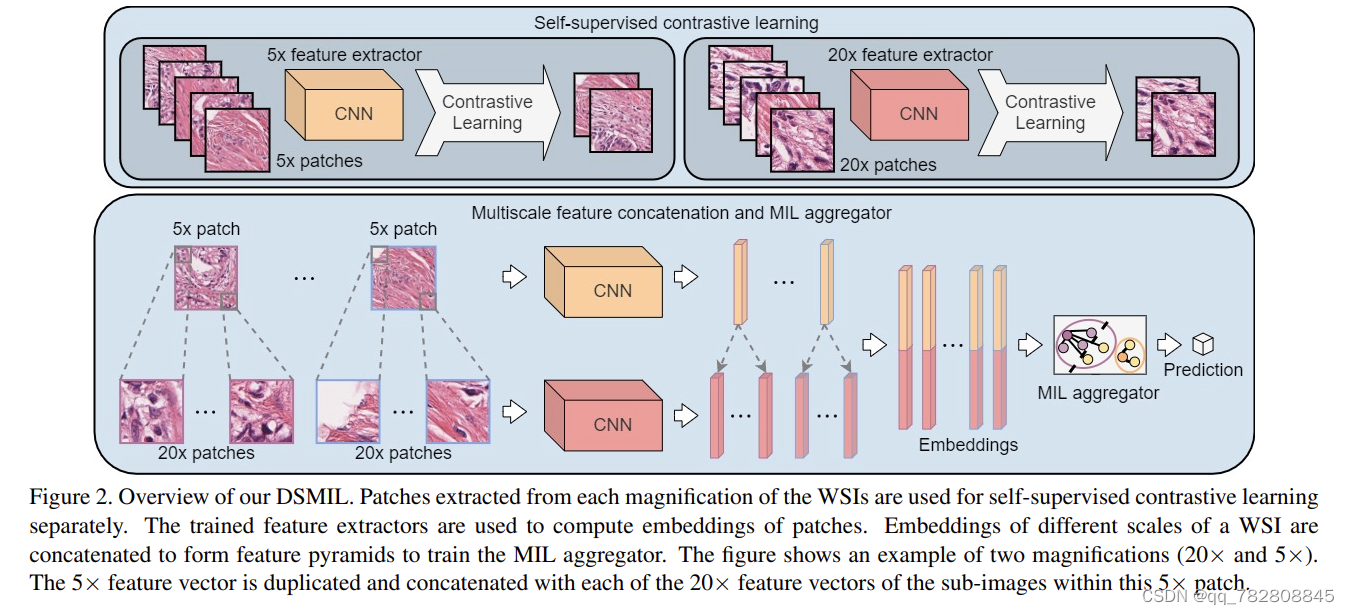
Dual-stream multiple instance learning network for whole slide image classification with self-supervised contrastive learning
这是CVPR2021的文章
看结构图就很一目了然,就是利用了多个分辨率的图像。获取了更多不同大小的视野。
基于Transformer的WSI分类模型
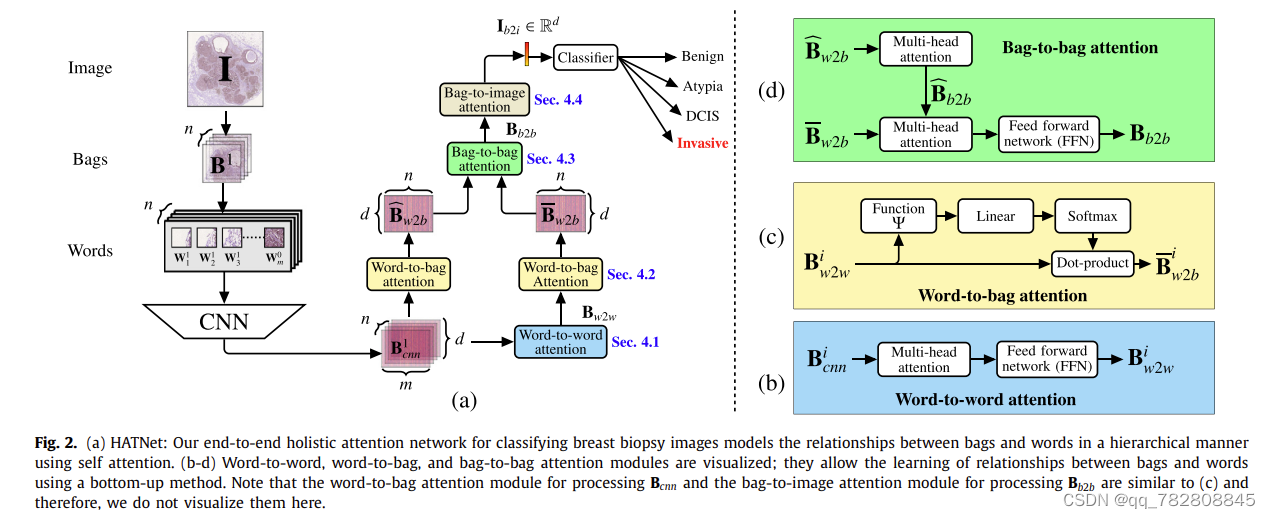
End-to-End diagnosis of breast biopsy images with transformers
MIA 2022的文章
从结构图中也能看出,其实就是Transformer的魔改。
TransMIL: Transformer based Correlated Multiple Instance Learning for Whole Slide Image Classification
NIPS2021的文章。算是比较有意思的WSI分类模型。
作者将每个patch作为一个词向量,输入Transformer,并且中间结合了卷积。效果很不错。
Data-efficient and weakly supervised computational pathology on whole-slide images
这个可能是现在follow最多的文章之一。CLAM。
就是patch级别特征的多实例学习。
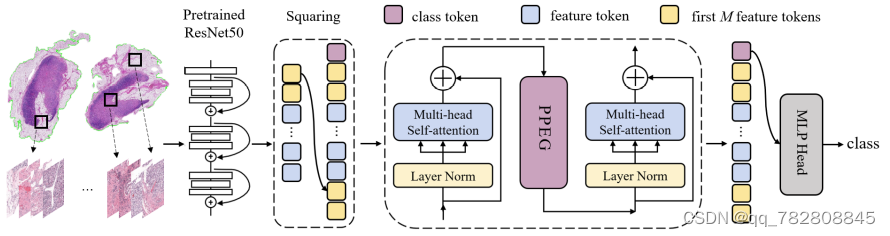
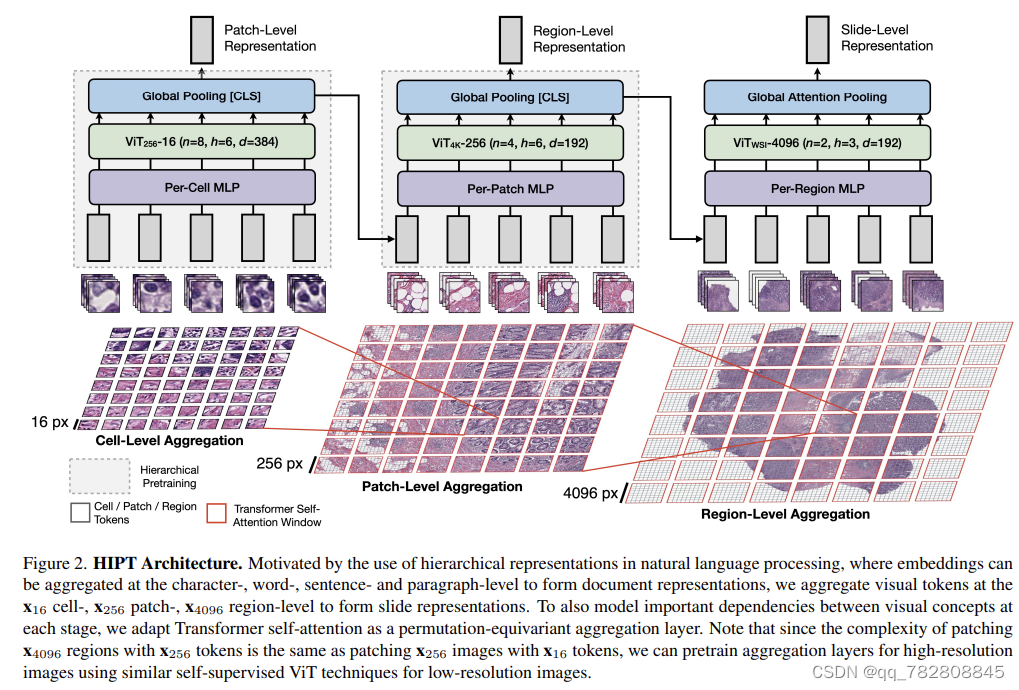
Scaling Vision Transformers to Gigapixel Images via Hierarchical Self-Supervised Learning
这是CVPR2022的文章。值得一提的是这也是CLAM这个文章同一个单位出的。他们对病理图像有着很不错的研究,大杂志发了很多。
当时一看这个图,我就知道这个文章是怎么做的。简直make sense到不行。就是把小倍率的patch送入transformer,然后将他的class token一级一级的往上聚合。非常符合WSI的存储方式,也符合医生的阅片习惯。
今天就介绍这么多吧。
2024最新激活全家桶教程,稳定运行到2099年,请移步至置顶文章:https://sigusoft.com/99576.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请联系我们举报,一经查实,本站将立刻删除。 文章由激活谷谷主-小谷整理,转载请注明出处:https://sigusoft.com/117492.html